导语:随着科技的不断发展,肿瘤的诊断和治疗也在不断地进步。其中,ctDNA甲基化检测在肿瘤临床诊疗中的应用越来越受到关注。本文将为大家介绍甲基化的原理以及甲基化检测在肿瘤诊疗全程中的作用。
一、DNA甲基化的化学本质
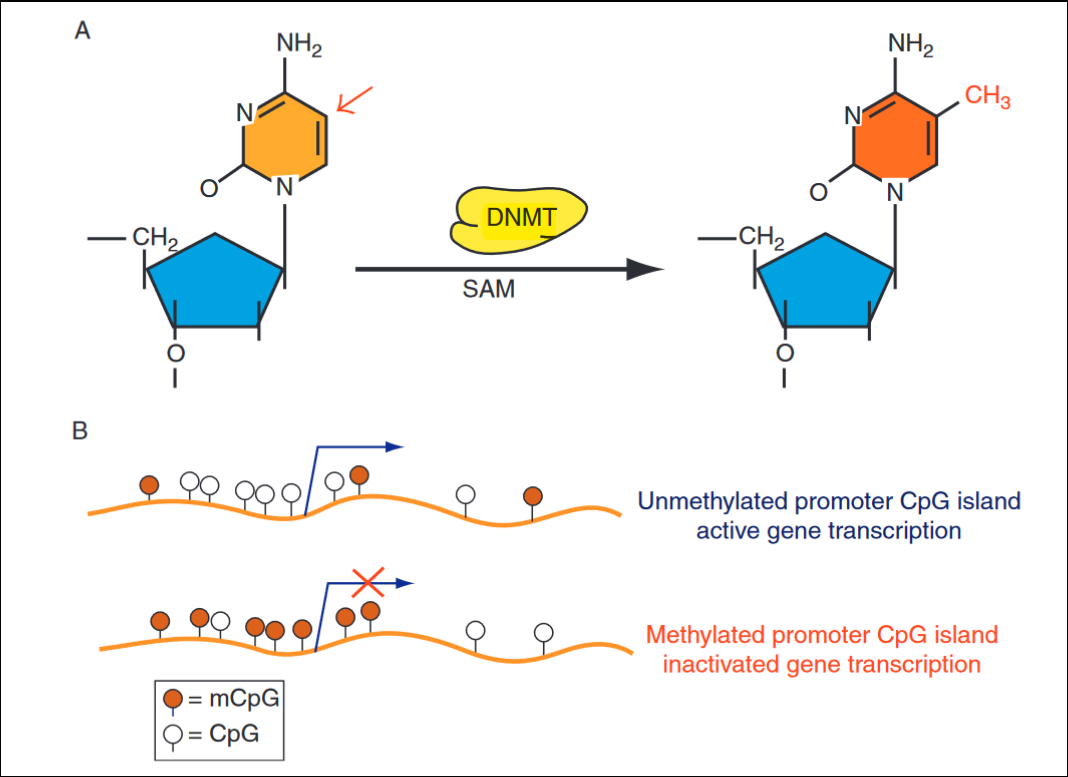
DNA甲基化是哺乳动物中研究的最深入的表观遗传修饰之一,其本质是在甲基转移酶(DNA methyltransferases, DNMTs)的作用下,胞嘧啶的第五位C原子上的氢被甲基基团取代,形成5’-甲基胞嘧啶(如图1A)。在人类基因组中大约有2800万个CpG位点,在正常体细胞中70%是甲基化状态。但DNA甲基化并不是均匀分布在基因组中的,有些分散于DNA序列中;另一些呈现高度聚集状态,称之为CpG岛,它们常位于转录调控区附近,与56%的人类基因组编码基因相关。
图1. DNA甲基化的化学本质¹
二、为什么ctDNA甲基化可以作为癌症标记物
适当的DNA甲基化对人体正常的细胞发育和功能表达至关重要,这一过程中的任何异常都可能导致包括癌症在内的各种疾病。肿瘤细胞的甲基化特征与正常细胞存在明显不同,我们可以观察到全域低甲基化和局部高甲基化。一方面是总体甲基化CpG含量下降,导致基因组不稳定;另一方面,特定基因启动子的CpG岛出现超甲基化,使得基因转录被抑制,这是许多肿瘤细胞的关键标志(如图1B)²。甲基化和肿瘤发生发展过程密不可分,而且与基因突变的高度个体化和异质性不同,同一类型的肿瘤细胞往往表现出相似的DNA甲基化变化³。而研究发现细胞游离肿瘤DNA (cell-free tumor DNA, ctDNA)的甲基化模式与它们起源的细胞或组织一致⁴,这也就意味着我们可以通过检测患者外周血中肿瘤特异性DNA甲基化畸变来进行癌症早期诊断和预后监测。
三、如何检测甲基化
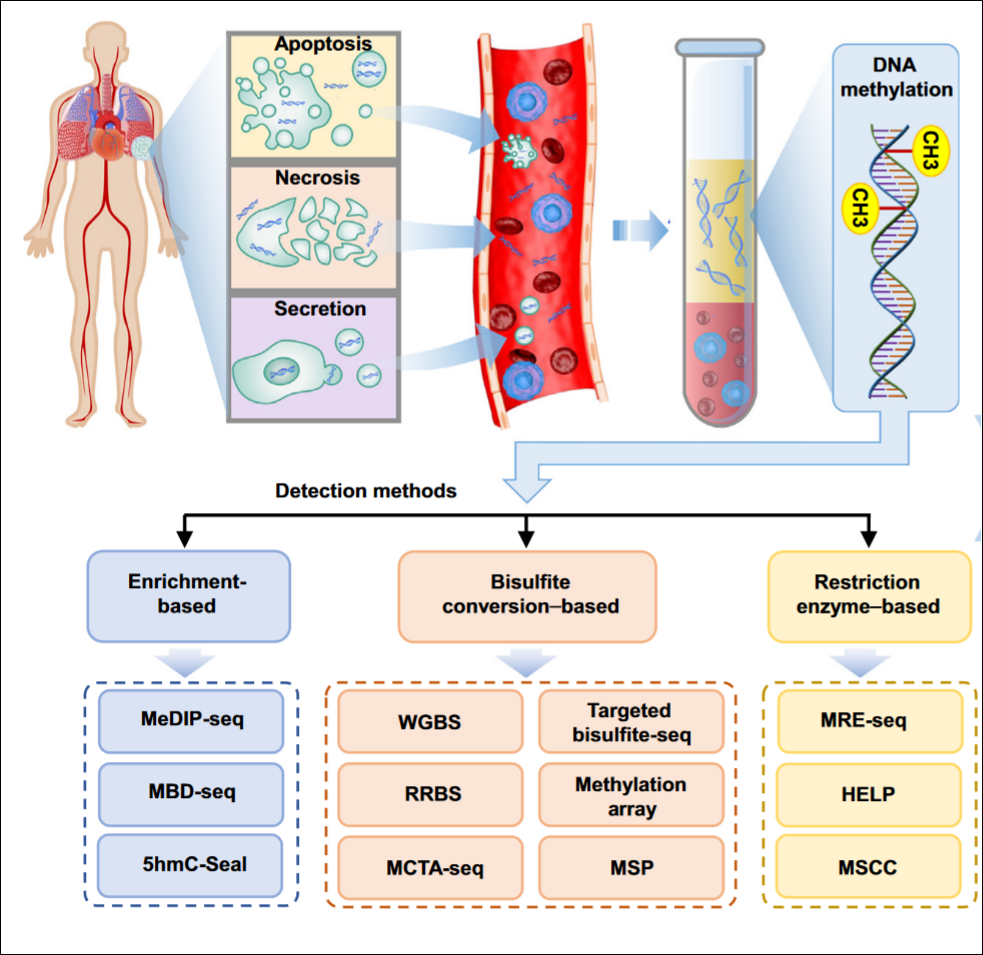
用于ctDNA甲基化标记的分析方法主要包括三类(如图2):
1)基于抗体富集处理的方法:甲基化DNA免疫沉淀测序(MeDIP-seq); 甲基CpG结合域蛋白捕获测序法(MBD)等;
2)基于亚硫酸氢盐处理的方法:包括多种甲基化特异性PCR(MSP、RT-MSP、ddMSP),全基因组甲基化测序(WGBS);焦磷酸测序等;
3)基于限制性内切酶处理的方法:HpaII-微小片段富集链接PCR(HELP); 甲基化限制性内切酶法(MRE-seq)等;
图2. DNA甲基化的检测方法⁵
四、ctDNA甲基化检测在肿瘤诊疗中的多样化临床应用
癌症筛查
DNA甲基化的改变发生在癌变的早期³,故甲基化ctDNA生物标志物是一种极具吸引力的癌症筛查方法,而且由于检测取样的非侵入性,可以有效提高癌症筛查的依从性。近年来,一些以游离细胞DNA(cell-free, cfDNA)甲基化为基础的生物标志物已被引入临床应用。最广为人知并且代表着甲基化的血液生物标志物从实验室走向临床实践的一个重要标志就是2016年美国FDA批准了首个基于血浆的SEPT9基因甲基化结直肠癌筛查方法Epi proColon。此外,美国早筛龙头Grail斥巨资开展了CCGA试验,利用甲基化特征对泛癌种进行早筛,另一家早筛企业Exact Sciences的Deep-C则是将甲基化与突变、蛋白等多组学结合,提高准确性来进行肠癌的早筛。
癌症早诊
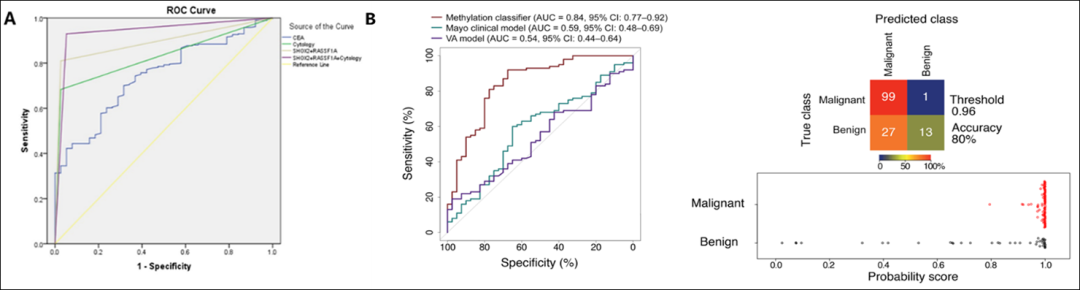
癌症的早期诊断对改善患者预后、降低病死率具有重要意义,但早期肿瘤体积小,很难以非侵入性方式将其与正常的生化变异区分开来。目前ctDNA甲基化标记的应用研究主要集中在癌症的早期诊断方面,一项研究在肺泡灌洗液中利用SHOX2和RASSF1A双基因甲基化检测对肺癌进行诊断,结果发现总体诊断效率显著高于细胞学检查和血清生物标志物癌胚抗原(如图3A)⁶。另一项研究⁷采用外周静脉血样本进行靶向DNA甲基化测序及深度学习方法,构建肺结节良恶性诊断模型PulmoSeek,用于早期肺癌的诊断效果也显著优于其他现有临床手段(如图3B)。目前,国内已获批的甲基化早期诊断试剂盒有17种,癌种主要以结直肠癌、肺癌为主,也有诸如宫颈癌、膀胱癌等其他癌种。普济生物自主研发的高通量数字PCR平台可实现对多个癌症相关甲基化基因的高灵敏度液体活检,对肺泡灌洗液中cfDNA的多基因甲基化检测模型显示其对肺癌诊断的敏感性高达93.3%,特异性100%。
图3. DNA甲基化检测在肿瘤早诊中的研究进展⁶⁷
MRD(Minimal residual disease)监测
手术或根治性治疗后的微小残留病灶(MRD)是肿瘤复发的主要原因。遗憾的是,多种肿瘤目前还没有有效的方法来识别MRD和复发。此外,用于复发风险分层的传统临床或病理参数不足以准确指导辅助治疗,这导致患者不得不接受毫无区别的治疗。只有将患者分层为高危组和低危组才能使我们能够向最有可能获益的患者提供辅助治疗,同时使低危患者避免不必要的合并症和不良事件风险。
一项前瞻性临床试验⁸评估了II-III期CRC患者术后ctDNA甲基化标志物(WIF1和NPY)与肿瘤复发的关系。研究在手术前后获得了805例II-III期结直肠癌患者的血浆样本并在术后第1次随访时采用数字微滴PCR检测ctDNA中WIF1和NPY的甲基化水平。结果,ctDNA阴性组的2年无病生存率显著高于ctDNA阳性组(82% vs 64%),提示ctDNA甲基化分析可用于预测术后残留的显微镜下病变。对87例II~III期乳腺癌患者进行的一项回顾性研究也报告了类似结果⁹。这表明,术后检测ctDNA中的肿瘤特异性甲基化也可以用于监测结直肠癌和其他癌症根治性治疗后的MRD。这种方法有可能成为癌症患者术后管理的关键工具。
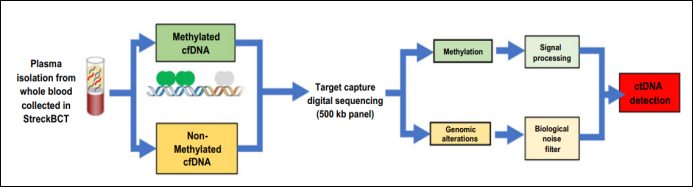
血检巨头Guardant Health在 Clinical Cancer Research披露了有关其获批的Guardant Reveal液体活检产品的详细研究数据,该产品在利用甲基化特征的同时联合了突变特征用于检测和监测早期结直肠癌患者(CRC,II和III期)的MRD(如图4),灵敏度达到91%,特异性达100%。Guardant Reveal是第一个利用ctDNA突变和ctDNA甲基化的MRD分析,通过他们的研究发现,首次界标采血时间(中位31.5天)的ctDNA突变MRD灵敏度仅有40.7%,ctDNA甲基化MRD敏度仅有48.2%,而两者结合的MRD灵敏度能提高到55.6%,这无疑是为MDR监测提供了新的思路¹⁰ 。
图4. Guardant Reveal利用ctDNA突变联合甲基化检测实现早期结直肠癌患者MRD监测
预测疗效和预后
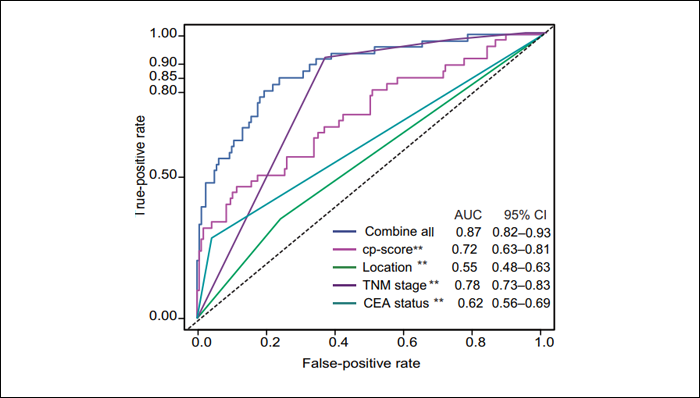
ctDNA的半衰期较短,这使其与肿瘤细胞状态直接相关,从而为治疗期间监测癌症负荷提供了连续动态观察的可能性¹¹。早在15年的ESMO大会上就报道了检测cfDNA中的MGMT高甲基化可预测多形性胶质母细胞瘤或转移性CRC患者接受细胞毒性烷化剂治疗后的反应和进展时间¹²。另一项研究在CRC患者中使用五种标记的cfDNA甲基化组合开发了一个预后模型。该预后模型的效能优于其他预后指标,如血清CEA状态、TNM分期或原发肿瘤位置。将这些常规预后标志物与cfDNA甲基化模型相结合可进一步提升预测效能¹³。
图5. 基于cfDNA甲基化和常规肿瘤标志物分析的结直肠癌生存预后预测性能¹³
ctDNA甲基化液体活检已经在癌症筛查、早诊、MRD监测、疗效预测及预后等方面崭露头角,然而,在其完全融入临床实践之前,还需要大量的多中心研究,以确定ctDNA甲基化检测的分析有效性和临床有效性。
参考文献(滑动):
[1] Kulis M, Esteller M. DNA methylation and cancer. Adv Genet. 2010;70:27-56. doi:10.1016/B978-0-12-380866-0.60002-2
[2] Paz MF, Fraga MF, Avila S, et al. A systematic profile of DNA methylation in human cancer cell lines. Cancer Res. 2003;63(5):1114-1121.
[3] Baylin SB, Jones PA. A decade of exploring the cancer epigenome - biological and translational implications. Nat Rev Cancer. 2011;11(10):726-734. doi:10.1038/nrc3130
[4] Moss J, Magenheim J, Neiman D, et al. Comprehensive human cell-type methylation atlas reveals origins of circulating cell-free DNA in health and disease. Nat Commun. 2018;9(1):5068. doi:10.1038/s41467-018-07466-6
[5] Luo H, Wei W, Ye Z, Zheng J, Xu RH. Liquid Biopsy of Methylation Biomarkers in Cell-Free DNA. Trends Mol Med. 2021;27(5):482-500. doi:10.1016/j.molmed.2020.12.011
[6] Zhang C, Yu W, Wang L, et al. DNA Methylation Analysis of the SHOX2 and RASSF1A Panel in Bronchoalveolar Lavage Fluid for Lung Cancer Diagnosis. J Cancer. 2017;8(17):3585-3591. doi:10.7150/jca.21368
[7] Liang W, Chen Z, Li C, et al. Accurate diagnosis of pulmonary nodules using a noninvasive DNA methylation test. J Clin Invest. 2021;131(10):e145973, 145973. doi:10.1172/JCI145973
[8] Taieb J, Taly V, Vernerey D, et al. LBA30_PR - Analysis of circulating tumour DNA (ctDNA) from patients enrolled in the IDEA-FRANCE phase III trial: Prognostic and predictive value for adjuvant treatment duration. Ann Oncol. 2019;30:v867. doi:10.1093/annonc/mdz394.019
[9] Takahashi H, Kagara N, Tanei T, et al. Correlation of Methylated Circulating Tumor DNA With Response to Neoadjuvant Chemotherapy in Breast Cancer Patients. Clin Breast Cancer. 2017;17(1):61-69.e3. doi:10.1016/j.clbc.2016.06.006
[10] Parikh AR, Van Seventer EE, Siravegna G, et al. Minimal Residual Disease Detection using a Plasma-only Circulating Tumor DNA Assay in Patients with Colorectal Cancer. Clin Cancer Res Off J Am Assoc Cancer Res. 2021;27(20):5586-5594. doi:10.1158/1078-0432.CCR-21-0410
[11] Tuaeva NO, Falzone L, Porozov YB, et al. Translational Application of Circulating DNA in Oncology: Review of the Last Decades Achievements. Cells. 2019;8(10):1251. doi:10.3390/cells8101251
[12] Barault L, Amatu A, Bleeker FE, et al. Digital PCR quantification of MGMT methylation refines prediction of clinical benefit from alkylating agents in glioblastoma and metastatic colorectal cancer. Ann Oncol Off J Eur Soc Med Oncol. 2015;26(9):1994-1999. doi:10.1093/annonc/mdv272
[13] Luo H, Zhao Q, Wei W, et al. Circulating tumor DNA methylation profiles enable early diagnosis, prognosis prediction, and screening for colorectal cancer. Sci Transl Med. 2020;12(524):eaax7533. doi:10.1126/scitranslmed.aax7533